SMART
アミノ酸配列を投入するとドメイン検索する。左下のpfamのチェックを忘れないように。画像のドメインをクリックするとそのアミノ酸配列とドメイン情報を表示する。
Sequence Manipulation Suite
配列操作のツール集。最初に左のリストからコマンドを選ぶ。EMBOSSに似ている。
DDBJ Read Annotation Pipeline
次世代シーケンスデータの解析ツール集。要アカウント。ログインしなくても使えるが、他者から解析内容が見えてしまうらしい。
ClustalW
核酸あるいはアミノ酸配列をアラインメントする。PCにTreeViewをインストールしておくと、dndファイルをダウンロードしたら自動的に系統樹を表示する。
BLAST
公共の配列データベースに対してホモロジー検索する。簡易系統樹を表示可能。
OPTIMIZER
アミノ酸配列から各種生物にコドンを最適化して塩基配列を作製する。指定した制限酵素配列を除外することも可能。
DNA to protein translation
DNA配列をペーストすると、ORFを表示する。それをクリックするとアミノ酸配列を表示する。配列をペーストした後、”reverse”および”complement”ボタンを押すと相補鎖に変換する。
PSORT
アミノ酸配列から細胞内局在を予想する。植物用、動物用などに分かれている。葉緑体、ミトコンドリア、パーオキシゾームなどに移行する確率をそれぞれ予想し、結果をそれぞれNegative or posiviteで表現する

TargetP
アミノ酸配列を投入すると細胞内局在を予想する。Chloroplast, Mitochondria, Secretaryもしくはそれ以外の4パターンのみ。

SignalP
アミノ酸配列を投入するとニューラルネットワークと隠れマルコフモデルによるシグナル配列部位をグラフで表示する。
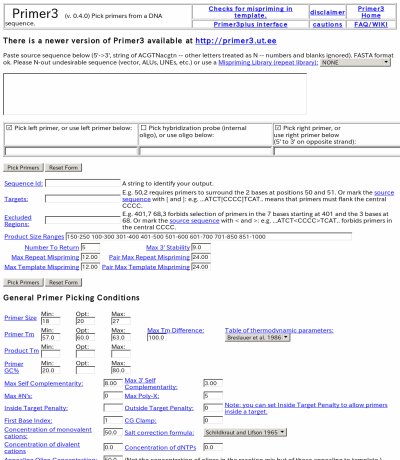
Primer3
DNA配列を投入するとPCRあるいはSequencing用のPrimerを示す。Primerの融点、長さ、GC含量、産物のサイズ、領域などこまかな設定が可能。数通りのPrimerを示す。Primerの位置をマップで示すのでわかりやすい。
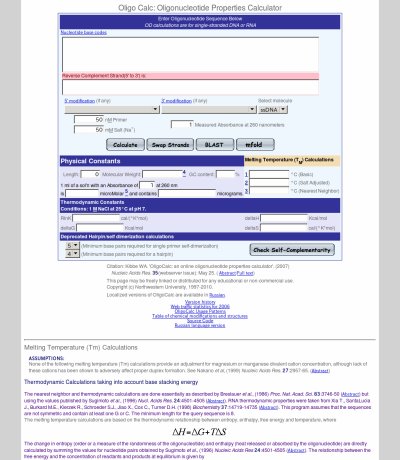
Oligo Calc
オリゴDNAの配列を投入すると Tmを表示する。
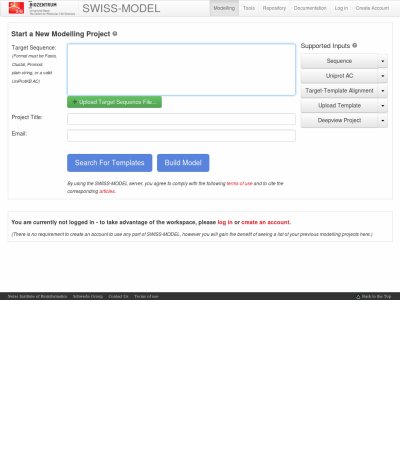
SWISS-MODEL
アミノ酸配列を投入すると立体構造データベースから類似タンパクを探して、その立体構造に基づいて自分のタンパクの立体構造を予測する。結果をPDB形式で出力し、emailで返送するので、RasMol、WebLab ViewerLiteなどで表示することができる。

BACPAC
BAC, PAC vectorを有償で分譲してくれる。BAC libraryなどもあり。

T-DNA Express: Arabidopsis Gene Mapping Tool
アラビドプシスの遺伝子名を入力すると、そこにタグが挿入されたクローンを地図で表示する。クローンは入手可能。
DotLet
2つのDNAやアミノ酸配列のドットマトリックス解析。"Input"ボタンを押して1つ目の配列を入力し、いったん窓を閉じて再び"Input"ボタンを押して2つ目の配列を入力。Horisontalとverticalの配列をそれぞれ指定して実行。結果をファイルに保存できない。要Java実行環境。
Compute pI/Mw tool
アミノ酸配列を投入すると、pIとMWを表示する。

Webcutter
塩基配列を投入すると制限酵素サイトを図とリストで表示する。

















